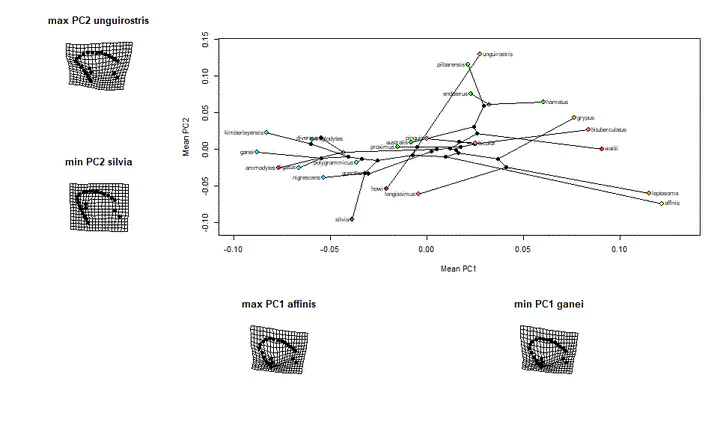
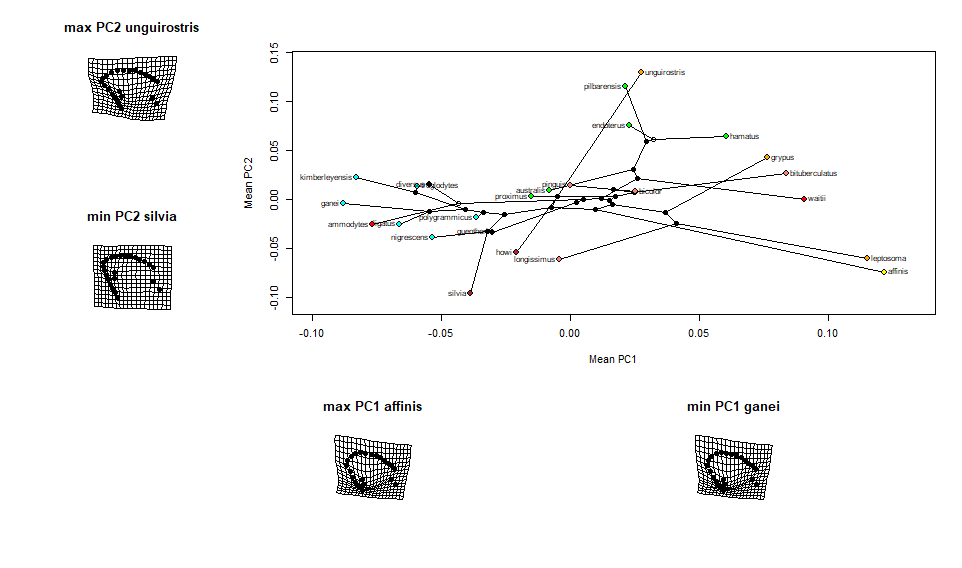
Phylomorphospace plot with relative values
Update Sept 2021: For the latest update please visit this Bitbucket repository
แปะไว้ให้ตัวเองจำ … วันหน้าอาจจะมาเขียน tutorial ดีๆ
เวลาจะแสดงความแตกต่างของลักษณะ geometric morphometrics ระหว่างสิ่งมีชีวิตหลากหลายสายพันธุ์ ข้อมูลที่สำคัญคือ:
- ความสัมพันธ์ระหว่างสายพันธุ์
- รูปลักษณะที่แตกต่างจากค่าเฉลี่ยของ species ที่ได้คะแนนสุดขั้ว
วันนี้นั่งวิเคราะห์งานอยู่จึงหาวิธีเขียน function ที่สามารถแสดงผลออกมาเป็น figure ได้ทันที อาจจะไม่สวยหรูแต่ช่วยได้เวลาจะเอาไปนำเสนอกับผู้ร่วมงานหรืออาจารย์ที่ปรึกษา
ถ้าใครสนใจ จะทำ figure อย่างในรูปขั้นต้นลองปรับ code ด้านล่างนี้ใช้นะครับ สิ่งสำคัญที่ต้องมีคือ
- species tree
- data frame ที่มี mean PC1 และ PC2 สำหรับแต่ละ species
- array ที่มี คะแนน landmark เฉลี่ยของทุกตัวอย่าง
Packages:
- geomorph
- phytools
R code for the anilios.phylomopho.maker() function
anilios.phylomopho.maker <- function(ypcs.df, means3d.df) {
# make a subset of mean pca scores that we have tips for head shape
Ypcs.trim <- ypcs.df %>%
semi_join(tree.tips, by = c('species' = 'species_in_tree'))
# prune tree again for only tips that we have data for
head_shape_df_tips <- as.vector(Ypcs.trim$species)
# PRUNED tree for head shape data
Ypcs.trim.tree <- sub_mt_tree %>%
keep.tip(., head_shape_df_tips)
# PCA matrix for head shape
pca.headshape.matrix <- as.matrix(Ypcs.trim[c("mean_PC1", "mean_PC2")])
rownames(pca.headshape.matrix) <- as.vector(Ypcs.trim$species)
# plot layouts
layout(matrix(c(1,2,2,
3,2,2,
4,5,6
), 3, 3, byrow = TRUE),
widths = c(1,1,2), heights = c(1,1,1))
# plot extreme values
means3d <- means3d.df
Ypcs <- as.matrix(ypcs.df)
# PC2max
plotRefToTarget(mshape(means3d),
mshape(means3d[,,which(Ypcs[,2]==max(Ypcs[,2]))]))
title(main = paste("max PC2", names(which(pca.headshape.matrix[,2]==max(pca.headshape.matrix[,2])))))
# plot phylomorphospace of head shape data
phylo_plot <- phylomorphospace(Ypcs.trim.tree, pca.headshape.matrix, label = "horizontal",
fsize = 1,
xlab = "Mean PC1", ylab = "Mean PC2",
control = list(col.node=color.all))
# PC2 min
plotRefToTarget(mshape(means3d),
mshape(means3d[,,which(Ypcs[,2]==min(Ypcs[,2]))]))
title(main = paste("min PC2", names(which(pca.headshape.matrix[,2]==min(pca.headshape.matrix[,2])))))
plot.new()
# PC1 max
plotRefToTarget(mshape(means3d),
mshape(means3d[,,which(pca.headshape.matrix[,1]==max(pca.headshape.matrix[,1]))]))
title(main = paste("max PC1", names(which(pca.headshape.matrix[,1]==max(pca.headshape.matrix[,1])))))
# PC1 min
plotRefToTarget(mshape(means3d),
mshape(means3d[,,which(Ypcs[,1]==min(Ypcs[,1]))]))
title(main = paste("min PC1", names(which(pca.headshape.matrix[,1]==min(pca.headshape.matrix[,1])))))
}
# Plot the phylomorphospace
anilios.phylomopho.maker(ypcs.df = Ypcs.dorsal.df, means3d.df = means3d.dorsal)